Arrivato a questo punto avrei volentieri raccontato la fase successiva del nostro esperimento, cioè avrei raccontato la mia esperienza di utilizzo di un sistema di interpretazione dei dati e di elaborazione dell'albero genetico delle probabili linee evolutive.
Mi tocca invece riconsocere che, a causa della mia ignoranza, nella strada fatta fin qui ho fatto un po' troppo i conti senza l'oste. E l'oste in questo caso è rappresentato dall'aver configurato un sistema con troppi gradi di libertà. In realtà le mutazioni ammesse non sono "tutte" quelle matematicamente possibili ma un solo sottoinsieme.
Senza addentrarmi in particolari tecnici della chimica organica, di cui non so nulla, occorre che consideriamo di dover modificare il nostro mondo virtuale in base a questi due ulteriori e inevitabili vincoli:
- I nucleotidi non sono da considerarsi separati a uno a uno ma raggruppati a tre a tre. Un gruppo di tre nucleotidi, l'unità minima da considerare, è detto Codone. Sono i codoni gli elementi da considerare per interpretare le informazioni genetiche (pur con le ulteriori specifiche che cercherò di fornire nel seguito).
- Non tutte le sequenze di nucleotidi sono ammesse; una volta raggruppati i nucleotidi in codoni, infatti, esistono alcuni codoni, i cosiddetti "Codoni di stop", che, dal punto di vista biologico hanno un (meta-)significato di "comando di interruzione della codifica dell'amminoacido" mentre, dal nostro punto di vista meramente computazionale, devono essere considerati alla stregua di "reserverd words"
Trovo a questo punto necessario, caro lettore, condividere questi due punti:
- Il resto del presente articolo conterrà soltanto informazioni sul concetto di codone, per lo più rintracciate in internet e in particolar modo su Wikipedia.
- Questo articolo costituisce una divagazione sul tema e si può saltare direttamente all'articolo successivo (quando lo pubblicherò...) senza pregiudicare la comprensione del filo logico; almeno senza pregiudicarla in modo più grave di quanto già non avvenga basandosi sulle mie sole capacità espressive...
L'oste conta a tre a tre
La versione per la quale ogni nucleotide contiene un pezzo di informazione genetica atto a determinare una espressione (=sintesi) di un amminoacido, che a sua volta è l'elemento costitutivo delle proteine, è una versione troppo semplicistica, ahimé. Senza addentrarci in dettagli tecnici, in cui, già al primo passo, mi perderei, limitiamoci ad una visione di tipo computazionale.
In effetti la vera regola di "computazione" (utilizzata, questo sì, da ogni organismo vivente noto su questo pianeta...) consiste nel considerare i nucleotidi a tre a tre e per ognuna di queste terne è univocamente determinato l'amminoacido che verrà sintetizzato.
Quindi nell'ipotetica successione:
ATGGTTACCAAA
occorre innanzitutto considerare di spezzare la successione in parti da tre elementi l'una come:
ATG-GTT-ACC-AAA
e grazie a una tabella fissa e deterministica di "conversioni", è possibile ottenere "in output" la seguente successione di amminoacidi che verrebbero sintetizzati:
(ATG=>)Metionina - (GTT=>)Valina - (ACC=>)Treolina - (AAA=>)Lisina
Questo fatto ci impone di riconsiderare la versione iniziale del nostro albero, in cui la catena di nucleotidi dovrà comunque essere fatta da un numero multiplo di 3 di basi (il che, per caso, si verificava già). Inoltre, nella nostra ipotesi per cui nell'articolo precedente ipotizzavamo in astratto di avere tutti elementi T che convertivamo in "bianchi", ora avremmo elementi TTT che sintetizzano Fenilalanina.
Sinonimia dei codoni e codoni di stop
Dal momento che parliamo di terne di valori su quattro possibili elementi, è facile convincersi che le combinazioni possibili sono  . Gli amminoacidi, tuttavia, non sono così numerosi, ma sono soltanto 20 (o 22 a seconda che si contino anche Selenocisteina e Pirrolisina, codificati, a partire da Codoni di stop, in casi particolari). Per i nostri fini, considerare che siano 20 sarà un'approssimazione più che accettabile.
. Gli amminoacidi, tuttavia, non sono così numerosi, ma sono soltanto 20 (o 22 a seconda che si contino anche Selenocisteina e Pirrolisina, codificati, a partire da Codoni di stop, in casi particolari). Per i nostri fini, considerare che siano 20 sarà un'approssimazione più che accettabile.
Che cosa possiamo dedurre da questa differenza di cardinalità di insiemi, fra quanto combinatoriamente sarebbe possibile e quanto invece è realizzato in natura? In termini matematici, possiamo dire che la funzione 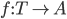 che mappa l'insieme delle terne di nucleotidi
che mappa l'insieme delle terne di nucleotidi  verso i 20 amminoacidi (insieme
verso i 20 amminoacidi (insieme 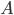 ) non è iniettiva.
) non è iniettiva.
La definizione usata invece dai biologi, per altro perfettamente equivalente, usa invece il concetto di sinonimia. In particolare, dato che, ad esempio, le seguenti terne:
- TTA
- TTG
- CTT
- CTC
- CTA
- CTG
codificano tutte per la Leucina, i biologi descriveranno queste terne come Sinonimi. Il che comporta, ad esempio, la considerazione seguente:
una modifica genetica che cambia il terzo nucleotide nel codone TTA e lo fa diventare TTG, produce un sinonimo del codone precedente perché, a livello della sua successiva "interpretazione", l'amminoacido sintetizzato sarà comunque la Leucina.
Viceversa:
se la modifica fosse stata da TTA verso, ad esempio, TTC, il codone ottenuto non sarebbe stato sinonimo dato che il codone TTC sintetizza la Fenilalanina.
Va da sé che le uniche variazioni che possono avere effetto (in positivo o in negativo) sull'espressione finale del fenotipo, sono quelle non-sinonimiche mentre le mutazioni sinonimiche non hanno alcun effetto sugli amminoacidi e conseguentemente sulle proteine prodotte e conseguentemente sul fenotipo prodotto e conseguentemente sulla eventuale fitness dell'organismo e conseguentemente sulla possibilità che l'organismo venga avvantaggiato o svantaggiato in termini di selezione naturale.
Riattribuiamo quadrati e colori
In questi termini appare più efficace, ai fini della pura rappresentazione del nostro mondo virtuale, ridefinire gli elementi rappresentativi, sia in termini di che cosa rappresentare sia in termini di come rappresentarlo.
Le variazioni che apporterò, per meglio recepire quanto sopra espresso, saranno:
- ridefiniamo il quadrato dalla versione
 quadratini, ognuno dei quali contiene una sequenza di 6 nucleotidi, alla versione a
quadratini, ognuno dei quali contiene una sequenza di 6 nucleotidi, alla versione a  quadratini, ognuno dei quali contiene un codone, quindi 3 nucleotidi.
quadratini, ognuno dei quali contiene un codone, quindi 3 nucleotidi. - ridefiniamo la modalità di colorazione associandola all'amminoacido codificato, secondo una tabella di conversione arbitraria (nel senso che non c'è una ragione particolare per associare a un Amminoacido il bianco piuttosto che il verde o il blu). In questo modo non solo i colori risultati possibili saranno solo tanti quanti gli amminoacidi codificati, cioè 20 (contro i precedenti
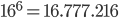 ) ma si otterrà anche una rappresentazione grafica più immediata del concetto di sinonimia fra codoni: due codoni differenti ma che codifichino per lo stesso amminoacido saranno rappresentati (cioè colorati) nello stesso modo.
) ma si otterrà anche una rappresentazione grafica più immediata del concetto di sinonimia fra codoni: due codoni differenti ma che codifichino per lo stesso amminoacido saranno rappresentati (cioè colorati) nello stesso modo.
Resterà invece invariata la lunghezza della sequenza complessiva di nucleotidi, che sarà di 1.728 nucleotidi, rappresentanti di 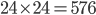 codoni.
codoni.
I Codoni di Stop
Un'altra differenza, magari con effetti limitati dal punto di vista grafico, ma significativa nella sostanza, sarà la necessità di dover escludere, fra le combinazioni possibili, gli eventuali Codoni di Stop, cioè le tre sequenze non ammesse in un codone in quanto sequenze che codificano, chimicamente parlando, l'informazione: "Interrompi espressione di amminoacidi". Questi codoni, che devono essere esclusi in quanto una catena di questo genere non avrebbe senso, sono tra l'altro escluse come possibilità e segnalate come errore anche dai programmi di analisi computazionale.
Con queste rettifiche siamo ora pronti per riprendere il cammino e reinventare la nostra evoluzione. Potremo tenere buona l'ipotesi di speciazione fatta nel grafo presentato in un articolo precedente e l'ipotesi di partire da un arbitraria successione di 1.728 T, pari a 576 Codoni che codifichino tutti Fenilalanina. Ma tutto il resto occorre riconsiderarlo, come faremo nel prossimo articolo.
